隨著針對許多細菌病原體的有效抗生素的逐漸喪失,噬菌體療法作為治療感染的手段引起了人們的新興趣�。一些噬菌體產(chǎn)生抗菌蛋白,導(dǎo)致其宿主細胞裂解����。研究人員最近確定了一種“YES”復(fù)合物的結(jié)構(gòu)�����,由噬菌體蛋白E和參與細胞壁生物合成的兩種蛋白質(zhì)MraY和SlyD組成�����。蛋白E阻斷MraY中的底物結(jié)合位點���,從而阻斷其必需的酶活性。這些發(fā)現(xiàn)可能有助于更好地了解噬菌體機制并助力噬菌體策略的開發(fā)�����。
作為其生命周期的一部分��,病毒必須從宿主中逃逸���。對于噬菌體�,這個過程需要突破細菌細胞壁和肽聚糖層�。小的單鏈DNA和RNA噬菌體已經(jīng)進化出單基因裂解蛋白,這些裂解蛋白會破壞肽聚糖的生物合成并觸發(fā)細胞裂解��,這是一些抗生素也使用的機制���。其中研究得最好的是來自歷史上重要的噬菌體ΦX174的蛋白質(zhì)E���,這是一種91個氨基酸的蛋白質(zhì),含有保守的N端跨膜螺旋和延伸的細胞質(zhì)C末端�����。
突變研究表明����,蛋白E
跨膜結(jié)構(gòu)域足以裂解,有功能的突變體映射到單個跨膜螺旋的一個面����。在蛋白E的這個結(jié)構(gòu)域中,保守的脯氨酸表現(xiàn)出最強的表型����,其中一種脯氨酸是裂解所必需的。野生型蛋白E需要組成型表達的細菌伴侶蛋白SlyD(對裂解D敏感)�,該蛋白包含脯氨酰異構(gòu)酶結(jié)構(gòu)域和伴侶結(jié)構(gòu)域。之前的工作已經(jīng)在MraY中發(fā)現(xiàn)了使其對蛋白e介導(dǎo)的溶解產(chǎn)生抗性的突變體��。MraY催化可溶性核苷-糖-肽和磷脂底物合成肽聚糖前體。雖然關(guān)于MraY還有很多功能上的問題��,但研究人員試圖了解噬菌體蛋白如何特異性地抑制這一關(guān)鍵酶�,以及SlyD
在這一過程中發(fā)揮了什么作用。
ΦX174從其細菌宿主中逃逸
研究人員建立了噬菌體蛋白E
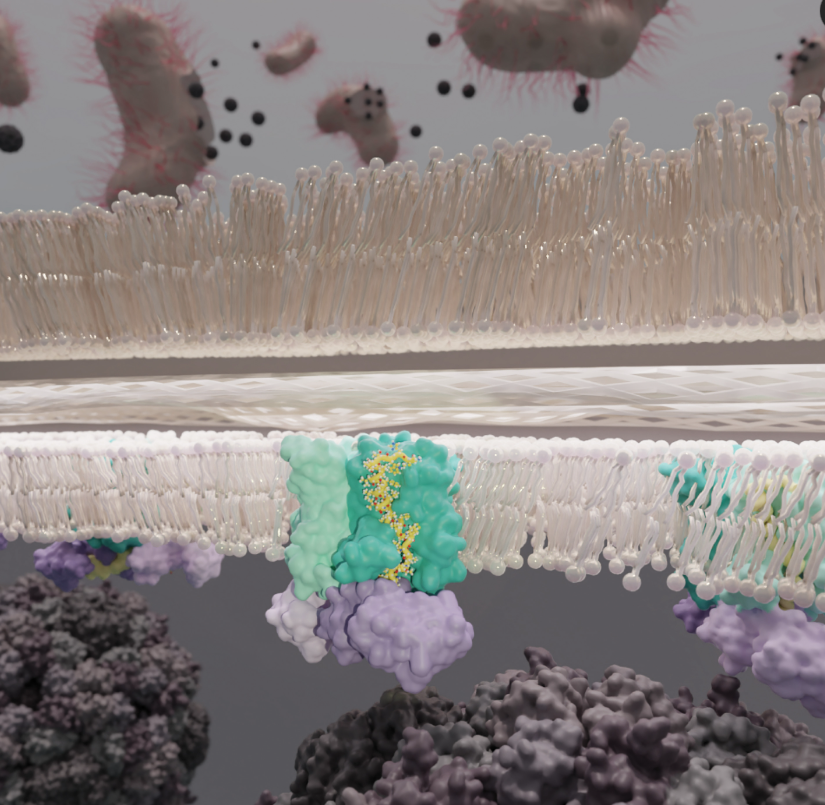
與大腸桿菌蛋白MraY和SlyD形成穩(wěn)定的復(fù)合體����,命名為YES復(fù)合體。在洗滌劑中溶解后�,通過單粒子電子冷凍顯微鏡確定了YES復(fù)合物的結(jié)構(gòu)——MraY二聚體采用背靠背的方向,暴露在膜上的活性位點彼此背對�����。兩個sld分子可以放入細胞質(zhì)側(cè)的密度中�����。兩條MraY和sld鏈由兩個蛋白E分子連接�����。蛋白E的跨膜螺旋在MraY上占據(jù)一個凹槽����,對應(yīng)于脂質(zhì)底物的假定結(jié)合位點。在細胞質(zhì)表面,蛋白E的跨膜結(jié)構(gòu)域發(fā)生彎曲���,穿過活性位點,然后形成一個α-螺旋��,延伸到sld的伴侶結(jié)構(gòu)域���。蛋白質(zhì)E的c末端繼續(xù)通過第二個sld中的一個肽結(jié)合槽并進入脯氨酰異構(gòu)酶活性位點����。其結(jié)果是蛋白E通過阻斷脂質(zhì)進入活性部位來抑制MraY��。之前的蛋白E突變表型可以通過包含必需的脯氨酸的結(jié)構(gòu)來解釋���,脯氨酸允許跨膜螺旋發(fā)生扭結(jié)���。對于MraY,我們可以解析包括TM1和TM2之間的保守環(huán)在內(nèi)的整個鏈�。我們證明了MraY的N端與TM2形成一個α-螺旋堆疊,并鑒定了蛋白質(zhì)表面有序的脂質(zhì)����。最后,我們提供了證據(jù)表明SlyD的作用是穩(wěn)定復(fù)合物。
蛋白E
通過阻斷MraY活性位點與SlyD形成穩(wěn)定復(fù)合物直接抑制MraY����。這一結(jié)構(gòu)解決了模型噬菌體ΦX174如何殺死細菌并逃逸細胞的關(guān)鍵問題。在ΦX174基因組中��,編碼蛋白質(zhì)E的基因在進化上受到其嵌入的基因D的限制�����。YES復(fù)合物為蛋白E的理性設(shè)計提供了一條超出基因D限制的途徑��。單基因裂解蛋白�,如蛋白E,為開發(fā)抗菌療法提供了有用的模型�。
原文:
The historically important phage ΦX174 kills its host bacteria by encoding a 91-residue protein antibiotic called protein E. Using single-particle electron cryo–microscopy, we demonstrate that protein E bridges two bacterial proteins to form the transmembrane YES complex [MraY, protein E, sensitivity to lysis D (SlyD)]. Protein E inhibits peptidoglycan biosynthesis by obstructing the MraY active site leading to loss of lipid I production. We experimentally validate this result for two different viral species, providing a clear model for bacterial lysis and unifying previous experimental data. Additionally, we characterize the Escherichia coli MraY structure—revealing features of this essential enzyme—and the structure of the chaperone SlyD bound to a protein. Our structures provide insights into the mechanism of phage-mediated lysis and for structure-based design of phage therapeutics.